2020
17
Genome editing in frumento duro
Arianna Frittelli è la vincitrice dell'AgroInnovation Award 2019 nella categoria "Innovazione varietale e genomica"

AgroInnovation Award è il premio di laurea al fine di promuovere la diffusione di approcci innovativi, strumenti digitali e l'utilizzo di internet in agricoltura
Secondo recenti stime dell'Organizzazione mondiale della sanità (Who), la malnutrizione dovuta alla carenza di nutrienti colpisce circa 2 miliardi di persone al mondo soprattutto nei paesi sottosviluppati e in via di sviluppo, portando a gravi conseguenze sanitarie, economiche e sociali. Dal momento che non è possibile risolvere il problema della malnutrizione attraverso la dieta, in quanto non è possibile assicurare una dieta equilibrata e variegata a tutta la popolazione mondiale, la biofortificazione delle colture di base (staple crops) dell'alimentazione rappresenta una valida soluzione.
Il frumento rappresenta la terza specie cerealicola più coltivata ed utilizzata al mondo. La sua adattabilità alla coltivazione, il buon profilo nutrizionale e la versatilità di trasformazione rappresentano le ragioni principali del suo successo, tuttavia rimane carente di micronutrienti come iodio (I), zinco (Zn), ferro (Fe) e vitamina A. Nonostante ad oggi si siano ottenuti buoni risultati nella biofortificazione di Zn e Fe, la biofortificazione di vitamina A rimane ancora poco affrontata.
La carenza di vitamina A (Vad, Vitamin a deficiency) rappresenta una delle maggiori cause di malnutrizione al mondo (Fig.1) e colpisce principalmente bambini e donne in gravidanza provocando danni alla vista che nel tempo possono portare a cecità, difficoltà di crescita e sviluppo dell'organismo, maggiore sensibilità verso le infezioni e malformazioni fetali.
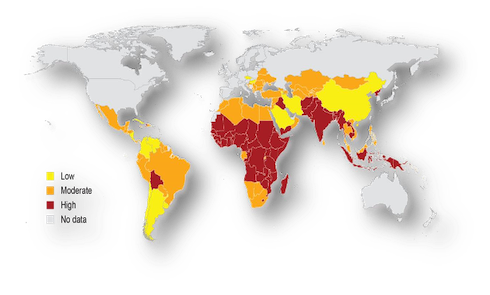
Fig.1 Diffusione della carenza di vitamina A
(Fonte: © Who)
Nello specifico la vitamina A, o retinolo, è una vitamina liposolubile presente in natura in diverse forme. Il retinolo come tale si trova negli alimenti di origine animale, mentre in quelli di origine vegetale si trovano i carotenoidi suoi precursori. I principali carotenoidi precursori della vitamina A sono β-carotene, β-criptoxantina e α-carotene; mentre i carotenoidi non provitamina A sono licopene, luteina e zeaxantina (Fig. 2).
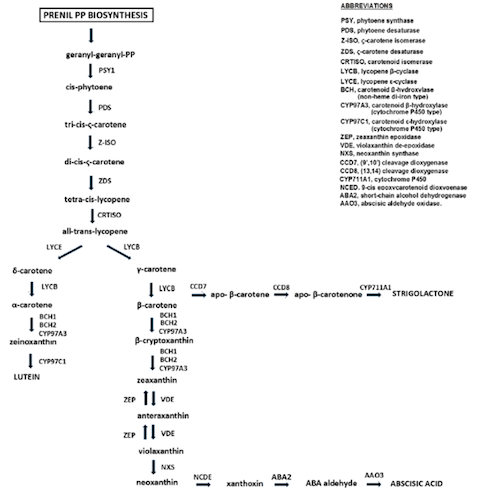
Fig. 2 Via biosintetica dei carotenoidi nelle piante superiori
(Fonte: © Colasuonno et al., 2017)
All'interno dei semi di frumento duro i carotenoidi si accumulano in basse quantità e sono principalmente carotenoidi non provitamina A, ma sono importanti per la colorazione della cariosside matura. Quest'ultimo aspetto, correlato con la colorazione di semole derivate dalla macinazione del frumento, ha un riscontro importante nella colorazione del prodotto finale e nella sua qualità nutrizionale. È necessario tenere in considerazione che il colore finale del prodotto si modifica durante lo stoccaggio e la lavorazione per via della degradazione ossidativa dei carotenoidi per mezzo di enzimi come perossidasi e lipossigenasi (Lpx).
L'obiettivo del lavoro è stato quello di sviluppare una strategia di ingegneria metabolica dei carotenoidi al fine di biofortificare in b-carotene le cariossidi di frumento duro attraverso il sistema CRISPR-Cas9.
In dettaglio, sono stati scelti come target tre geni codificanti tre enzimi chiave: l'epsilon licopene cicalsi (LCYE) e la b-carotene idrossilasi (BCH-1) coinvolti nella biosintesi dei carotenoidi e la lipossigenesi (Lpx-1), responsabile della degradazione ossidativa dei carotenoidi nelle fasi post raccolta.
Sulla base dell'analisi in silico dei geni target sono state disegnate sei guide a RNA (gRNA), due per ciascun gene target. Sono stati quindi assemblati i costrutti per la trasformazione genetica. Nello specifico sono stati utilizzati due costrutti: uno contenente la Cas9 e uno contenente le guide. Quest'ultimo è stato assemblato utilizzando una strategia multiplex che permette di bersagliare contemporaneamente i geni target sfruttando il sistema di processamento endogeno dei tRNA grazie al quale tutte le guide possono essere inserite all'interno di una struttura denominata PTG (polycistronic tRNA-gRNA). Il PTG nel momento in cui è trascritto è in grado di rilasciare i singoli gRNA pronti per indirizzare la Cas9 verso il sito di taglio bergaglio (Fig.3).
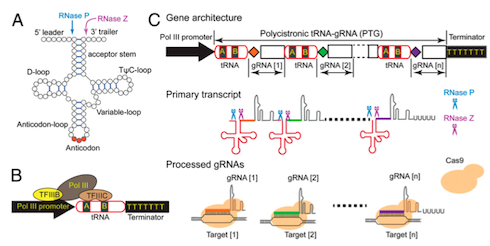
Fig. 3 Sistema multiplex per l'editing del genoma progettato sul sistema di processamento endogeno dei tRNA
(Fonte: © Xie et al., 2015)
I costrutti assemblati sono stati moltiplicati in cellule di Escherichia coli, quindi estratti e controllati tramite digestione e sequenziamento ed infine inseriti in pianta attraverso metodo biolistico. Le piante sono state rigenerate e sottoposte a screening per verificare l'avvenuta trasformazione.
La presenza dei costrutti nelle piante rigenerate T0 è stata verificata mediante PCR utilizzando coppie di primers disegnati direttamente sui vettori. Sulle piante in cui era stata verificata la presenza dei costrutti è stato condotto uno screening preliminare mediante PCR al fine di verificare la presenza di eventi di editing che abbiano causato grandi delezioni nei geni bersagliati. I profili elettroforetici delle analisi effettuate per i tre geni target non hanno mostrato differenze e grandi delezioni o inserzioni rispetto al wild-tipe tranne nel campione 87 per il gene Lpx-B1.2. Il campione è stato quindi sequenziato e la sequenza nucleotidica ha mostrato una delezione di 48 bp rispetto alla sequenza del wild-tipe che risulta cadere nella porzione della sequenza nucleotidica compresa tra le due gRNA scelte per l'editing confermando anche la funzionalità delle guide a RNA (Fig.4).
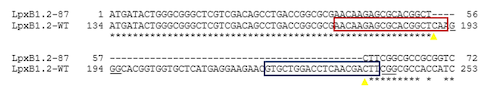
Fig. 4 Allineamento delle sequenze nucleotidiche per il gene Lpx-B1.2del campione 87 e del wild-type. Nei rettangoli sono indicate le guide a RNA disegnate sul gene; le sequenze sottolineate rappresentano le rispettive PAM dei gRNA; i triangoli gialli indicano il sito di taglio della Cas9
Le analisi elettroforetiche non hanno permesso di individuare piccole modifiche (sostituzione nucleotidiche e piccole mutazioni di tipo Indel), che potrebbero causare l'interruzione o il frameshift della trascrizione dei geni di interesse e quindi generare il fenotipo desiderato. Per tale ragione, sarà necessario analizzare la generazione T1 con altri metodi di indagine (sequenziamento diretto, HRM genotyping, NGS) e selezionare le piante mutanti per i geni d'interesse privi di costrutti.
Ringrazio il mio relatore Francesco Sestili, il mio correlatore Francesco Camerlengo e tutto il Laboratorio di genetica e biochimica delle proteine vegetali dell'Università degli Studi della Tuscia.

Arianna Frittelli, categoria "Innovazione varietale e genomica"
(Fonte foto: © Arianna Frittelli)
Scarica la tesi completa di Arianna Frittelli
Per eventuali contatti: a.frittelli@unitus.it
AgroInnovation Award è il premio di laurea istituito da Image Line in collaborazione con l'Accademia dei Georgofili al fine di promuovere la diffusione di approcci innovativi, strumenti digitali e l'utilizzo di internet in agricoltura.

Leggi le tesi vincitrici della terza edizione.
Il frumento rappresenta la terza specie cerealicola più coltivata ed utilizzata al mondo. La sua adattabilità alla coltivazione, il buon profilo nutrizionale e la versatilità di trasformazione rappresentano le ragioni principali del suo successo, tuttavia rimane carente di micronutrienti come iodio (I), zinco (Zn), ferro (Fe) e vitamina A. Nonostante ad oggi si siano ottenuti buoni risultati nella biofortificazione di Zn e Fe, la biofortificazione di vitamina A rimane ancora poco affrontata.
La carenza di vitamina A (Vad, Vitamin a deficiency) rappresenta una delle maggiori cause di malnutrizione al mondo (Fig.1) e colpisce principalmente bambini e donne in gravidanza provocando danni alla vista che nel tempo possono portare a cecità, difficoltà di crescita e sviluppo dell'organismo, maggiore sensibilità verso le infezioni e malformazioni fetali.

Fig.1 Diffusione della carenza di vitamina A
(Fonte: © Who)
Nello specifico la vitamina A, o retinolo, è una vitamina liposolubile presente in natura in diverse forme. Il retinolo come tale si trova negli alimenti di origine animale, mentre in quelli di origine vegetale si trovano i carotenoidi suoi precursori. I principali carotenoidi precursori della vitamina A sono β-carotene, β-criptoxantina e α-carotene; mentre i carotenoidi non provitamina A sono licopene, luteina e zeaxantina (Fig. 2).

Fig. 2 Via biosintetica dei carotenoidi nelle piante superiori
(Fonte: © Colasuonno et al., 2017)
All'interno dei semi di frumento duro i carotenoidi si accumulano in basse quantità e sono principalmente carotenoidi non provitamina A, ma sono importanti per la colorazione della cariosside matura. Quest'ultimo aspetto, correlato con la colorazione di semole derivate dalla macinazione del frumento, ha un riscontro importante nella colorazione del prodotto finale e nella sua qualità nutrizionale. È necessario tenere in considerazione che il colore finale del prodotto si modifica durante lo stoccaggio e la lavorazione per via della degradazione ossidativa dei carotenoidi per mezzo di enzimi come perossidasi e lipossigenasi (Lpx).
L'obiettivo del lavoro è stato quello di sviluppare una strategia di ingegneria metabolica dei carotenoidi al fine di biofortificare in b-carotene le cariossidi di frumento duro attraverso il sistema CRISPR-Cas9.
In dettaglio, sono stati scelti come target tre geni codificanti tre enzimi chiave: l'epsilon licopene cicalsi (LCYE) e la b-carotene idrossilasi (BCH-1) coinvolti nella biosintesi dei carotenoidi e la lipossigenesi (Lpx-1), responsabile della degradazione ossidativa dei carotenoidi nelle fasi post raccolta.
Sulla base dell'analisi in silico dei geni target sono state disegnate sei guide a RNA (gRNA), due per ciascun gene target. Sono stati quindi assemblati i costrutti per la trasformazione genetica. Nello specifico sono stati utilizzati due costrutti: uno contenente la Cas9 e uno contenente le guide. Quest'ultimo è stato assemblato utilizzando una strategia multiplex che permette di bersagliare contemporaneamente i geni target sfruttando il sistema di processamento endogeno dei tRNA grazie al quale tutte le guide possono essere inserite all'interno di una struttura denominata PTG (polycistronic tRNA-gRNA). Il PTG nel momento in cui è trascritto è in grado di rilasciare i singoli gRNA pronti per indirizzare la Cas9 verso il sito di taglio bergaglio (Fig.3).

Fig. 3 Sistema multiplex per l'editing del genoma progettato sul sistema di processamento endogeno dei tRNA
(Fonte: © Xie et al., 2015)
I costrutti assemblati sono stati moltiplicati in cellule di Escherichia coli, quindi estratti e controllati tramite digestione e sequenziamento ed infine inseriti in pianta attraverso metodo biolistico. Le piante sono state rigenerate e sottoposte a screening per verificare l'avvenuta trasformazione.
La presenza dei costrutti nelle piante rigenerate T0 è stata verificata mediante PCR utilizzando coppie di primers disegnati direttamente sui vettori. Sulle piante in cui era stata verificata la presenza dei costrutti è stato condotto uno screening preliminare mediante PCR al fine di verificare la presenza di eventi di editing che abbiano causato grandi delezioni nei geni bersagliati. I profili elettroforetici delle analisi effettuate per i tre geni target non hanno mostrato differenze e grandi delezioni o inserzioni rispetto al wild-tipe tranne nel campione 87 per il gene Lpx-B1.2. Il campione è stato quindi sequenziato e la sequenza nucleotidica ha mostrato una delezione di 48 bp rispetto alla sequenza del wild-tipe che risulta cadere nella porzione della sequenza nucleotidica compresa tra le due gRNA scelte per l'editing confermando anche la funzionalità delle guide a RNA (Fig.4).

Fig. 4 Allineamento delle sequenze nucleotidiche per il gene Lpx-B1.2del campione 87 e del wild-type. Nei rettangoli sono indicate le guide a RNA disegnate sul gene; le sequenze sottolineate rappresentano le rispettive PAM dei gRNA; i triangoli gialli indicano il sito di taglio della Cas9
Le analisi elettroforetiche non hanno permesso di individuare piccole modifiche (sostituzione nucleotidiche e piccole mutazioni di tipo Indel), che potrebbero causare l'interruzione o il frameshift della trascrizione dei geni di interesse e quindi generare il fenotipo desiderato. Per tale ragione, sarà necessario analizzare la generazione T1 con altri metodi di indagine (sequenziamento diretto, HRM genotyping, NGS) e selezionare le piante mutanti per i geni d'interesse privi di costrutti.
Ringrazio il mio relatore Francesco Sestili, il mio correlatore Francesco Camerlengo e tutto il Laboratorio di genetica e biochimica delle proteine vegetali dell'Università degli Studi della Tuscia.

Arianna Frittelli, categoria "Innovazione varietale e genomica"
(Fonte foto: © Arianna Frittelli)
Scarica la tesi completa di Arianna Frittelli
Per eventuali contatti: a.frittelli@unitus.it
AgroInnovation Award è il premio di laurea istituito da Image Line in collaborazione con l'Accademia dei Georgofili al fine di promuovere la diffusione di approcci innovativi, strumenti digitali e l'utilizzo di internet in agricoltura.

Leggi le tesi vincitrici della terza edizione.

